Dans le dossier consacré aux analyses d’ADN environnemental mis en ligne sur le site IBMA en avril 2015, les informations rassemblées indiquaient une certaine prééminence des travaux sur les animaux. Nous avions toutefois présenté certaines recherches portant sur les plantes aquatiques, dont les lentilles d’eau et diverses espèces invasives, issues d’investigations de chercheurs canadiens, chinois et européens. Ces travaux avaient tous pour principal objectif de déterminer les meilleurs marqueurs génétiques des espèces examinées à partir des plantes elles-mêmes, prélevées dans la nature ou achetées en jardineries et ne comportaient pas, à notre connaissance, de prélèvements d’eau en milieux naturels. Mais les chercheurs continuent de travailler…

Dans leur article paru en février 2016 dans la revue Freshwater Science, cinq chercheurs japonais ont présenté les résultats de leurs travaux portant sur la détection in situ d’ADN environnemental d’Egeria densa (Fujiwara et al., 2016). Cette espèce immergée d’Hydrocharitacée est considérée comme invasive au Japon où elle a été introduite au début du 20ième siècle comme espèce destinée à des recherches expérimentales. Sa dispersion ultérieure en milieu naturel lui a permis une naturalisation dans les années 40 et, depuis une très importante colonisation au début des années 80 du lac Biwa, un des plus grands lacs naturels du Japon, cette plante colonise de nombreux milieux aquatiques japonais, plans d’eau et cours d’eau (Figure 1).
Les recherches ont porté sur l’établissement d’une méthode spécifique de détection d’E. densa à l’aide de son ADN, puis sur la détection d’ADN environnemental (ADNe) à partir de prélèvements d’eau en conditions de laboratoire et en plans d’eau naturels.
Des amorces génétiques d’E. densa avaient déjà été proposées par Scriver et ses collègues mais la présence au Japon d’Elodea nuttallii, une autre Hydrocharitacée également invasive au Japon, a conduit les chercheurs japonais à utiliser des amorces pouvant discriminer ces deux espèces génétiquement identiques à 99 %.
Les essais en laboratoire ont consisté en des prélèvements d’eau étalés sur une période de 7 jours dans des aquariums contenant la même quantité de plantes (3 aquariums tests et un témoin sans plantes). Avec une périodicité des prélèvements très rapprochée durant le premier jour d’expérimentation puis plus longue par la suite, les résultats des analyses d’ADNe montrent un pic de détection après 24 à 48 H, puis une forte baisse à partir du cinquième jour dans deux aquariums sur trois (Figure 2). La forte concentration d’ADNe détectée dans le troisième aquarium le septième jour pourrait être liée au fait que cet aquarium contenait beaucoup plus de débris de plantes que les deux autres, débris dont la décomposition pourrait expliquer cette forte valeur.

Les prélèvements en milieux naturels ont été réalisés sur 23 plans d’eau situés dans la préfecture d’Hiroshima, sur lesquels, hormis leur positionnement GPS, aucune information n’est fournie (dimensions, qualité des eaux, peuplements végétaux, …). Ils sont dénommés “ponds” (étangs) dans l’article. Sur chaque plan d’eau, une observation de 10 minutes depuis les rives a permis de déterminer si E. densa y était présente. Un prélèvement d’un litre d’eau pour analyse ADNe a été réalisé avec des équipements (bouteille et entonnoir) javellisés au préalable pendant 10 minutes pour éviter toute contamination.
Les observations de terrain ont permis de détecter la présence d’E. densa dans 5 des 23 plans d’eau examinés. Cette présence a été confirmée par les analyses d’ADNe, avec pour quatre plans d’eau des résultats positifs pour les six analyses réalisées et deux sur six pour le cinquième. Aucune détection d’ADNe appartenant à E. densa n’a été faite dans les autres plans d’eau où E. densa n’a pas été observée.
Les auteurs en concluent donc que la détection de la présence en milieu naturel d’Egeria densa est possible à l’aide de l’analyse d’ADN environnemental. Ils remarquent que d’autres études sont nécessaires pour élargir cette approche à d’autres taxa et préciser l’utilité générale de cette technique de détection des plantes aquatiques. Ils rappellent également les intérêts que cette méthode leur semble présenter, entre autres dans :
- la facilitation de la détection des espèces en toutes saisons, quel que soit le type de milieu (observations directes quelquefois difficiles en raison de la turbidité des eaux ou de la profondeur d’implantation des espèces),
- la limitation des erreurs de détermination (dès lors que les traceurs génétiques spécifiques sont connus),
- la possibilité que ces travaux de détection puissent être réalisés par des personnes sans expertise taxonomique.
Ils terminent en indiquant que des progrès dans les méthodes d’analyse d’ADNe pourraient ultérieurement permettre des estimations de la biomasse des plantes (“further progress on eDNA analytical methods will enable us to estimate the biomass of plant species”).
Sur ce point, ils se réfèrent à deux articles datant de 2014 et 2015 portant sur la détection d’ADNe de la carpe commune et des résultats tentant de préciser des corrélations entre les concentrations en ADNe et l’abondance ou la biomasse des poissons, en milieu naturel dans le premier cas, en mésocosmes dans le second.
Il semble toutefois qu’ils soient plutôt optimistes dans leur extrapolation : si des progrès sont sans aucun doute à venir, il semble que des difficultés notables subsistent pour arriver à de telles estimations, et pas seulement parce qu’il s’agirait de passer des poissons aux plantes.
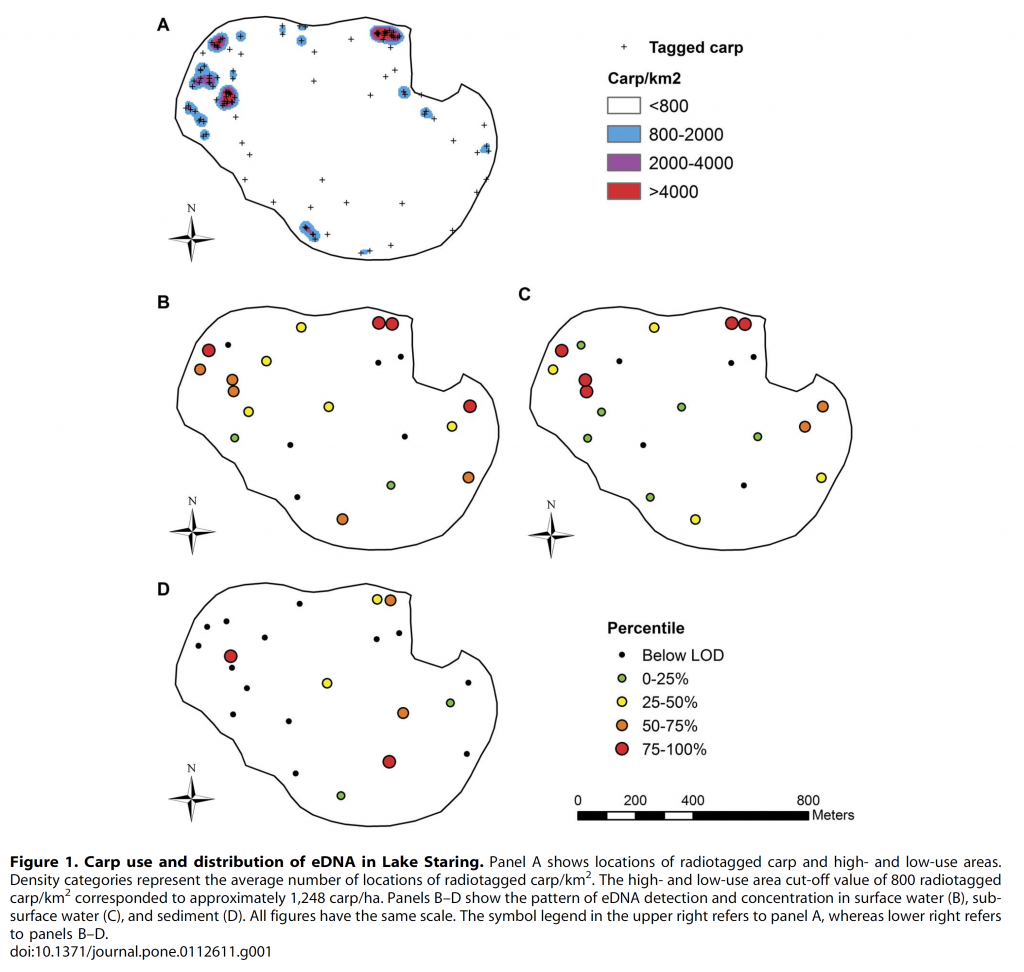
En effet, dans leurs conclusions sur leurs travaux dans un lac nord-américain d’environ 65 ha, Eichmiller et ses collègues (2014) remarquent entre autres choses que les mécanismes qui éliminent les fragments d’ADNe de la colonne d’eau, comme la décomposition et la sédimentation, sont rapides (“the mechanisms that remove eDNA from the water column, such as decay and sedimentation, are rapid”) et que les concentrations en ADNe sont extrêmement variables dans des échantillons éloignés de moins de 100 m (“eDNA concentration was shown to vary dramatically between samples taken less than 100 m apart”) (Figure 4).
Les travaux en mésocosmes de Doi et al. (2015) conduits sur des carpes introduites avec des effectifs différents dans des bassins de 450 L ont testé deux méthodes d’analyses d’ADNe, l’une déjà classique (qPCR), l’autre apparemment plus récente, permettant une quantification directe de l’ADN cible (DdPCR, “ droplet digital PCR”). Les auteurs constatent que les corrélations entre concentrations en ADNe et abondance des carpes dans les mésocosmes sont meilleures que celles avec la biomasse. Selon eux, cela peut s’expliquer par le fait que les poissons utilisés dans l’expérimentation étaient tous de même taille et que les quantités d’ADNe qu’ils pouvaient rejeter dans les eaux étaient similaires, induisant cette meilleure corrélation avec leur nombre. Cette uniformité des poissons ne se rencontre pas en milieu naturel. Les auteurs signalent également l’existence d’une nouvelle technique moléculaire, le séquençage à haut débit (“high-throughput sequencing”) et terminent en expliquant que la combinaison de plusieurs technologies pourrait permettre de meilleures méthodes d’évaluation de la répartition et de l’abondance et de la biomasse des espèces et des communautés (“By combining multiple technologies, we can develop better eDNA methods to evaluate the distribution and abundance/biomass of species and communities”).
Les données de concentration en “ADNe E.densa” figurant dans l’article de Fujiwara et al. (2016) permettent quelques commentaires sur des aspects plus directement liés aux plantes aquatiques.
La valeur très élevée (par rapport au corpus de données disponibles dans l’article) dans le troisième aquarium au bout de 7 jours d’expérimentation (environ 130 000 copies “ADN E. densa“/L) (figure 2), alors que les valeurs dans les deux autres aquariums restaient très faibles, a été attribuée par les auteurs au fait que la quantité de débris dans cet aquarium était élevée. La décomposition en cours de ces débris pourrait donc expliquer un relargage très important de fragments d’ADN dans l’eau environnante. L’état physiologique des plantes au moment des prélèvements d’eau dans le milieu prendrait alors une grande importance dans les interprétations qui pourraient être tirées des résultats des analyses ADNe : une concentration très élevée d’ADNe serait-elle l’indice d’une grande abondance ou biomasse de plantes présentes dans le milieu ou seulement d’une population de faible ampleur en voie de dégradation ?
Les concentrations moyennes en copies “ADN E. densa“/L obtenues dans les prélèvements d’eau en milieu naturel sont beaucoup plus faibles : elles varient entre environ 500 et 3800 pour les quatre plans d’eau où les six analyses d’eau se sont révélées positives et la valeur calculée pour le plan d’eau où seulement deux analyses sur six étaient positives n’est que de 12,1. Dans la mesure où l’article ne fournit qu’une simple indication de présence de la plante dans le plan d’eau, sans indication sur l’ampleur de la colonisation et que par ailleurs, un seul échantillon d’eau semble avoir été prélevé pour réaliser les six analyses d’ADNe, il n’est évidemment pas possible d’en tirer une quelconque hypothèse quant aux éventuelles différences de colonisation par E. densa entre ce plan d’eau et les quatre autres. Ce n’était pas l’objectif de ces travaux et, à l’instar de ce qui a par exemple été développé par Eichmiller et al. (2014) comme stratégie en lac pour les poissons, des recherches restent nécessaires pour définir des stratégies d’échantillonnages des milieux naturels pouvant permettre d’avancer dans la caractérisation des relations entre abondance des espèces cibles et les concentrations en ADNe.
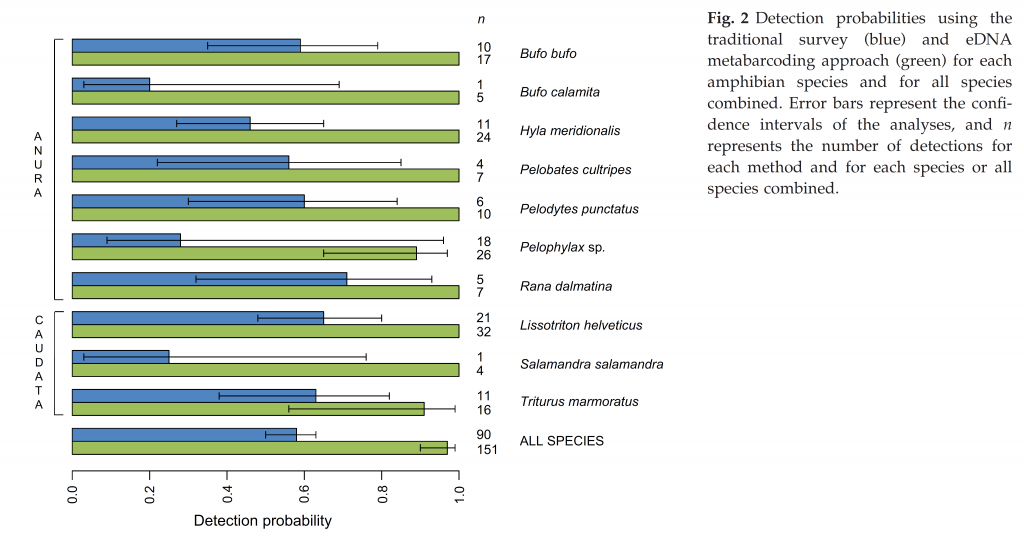
Il est indéniable que cette approche moléculaire présente de très grands intérêts dans la détection des espèces, dont les espèces invasives, et dans les inventaires de biodiversité qu’elle rend envisageable à des échelles spatiales très diverses et à des coûts plus réduits que ceux des méthodes actuelles d’inventaires. Comme le présente l’article très récent de Valentini et al. (2016), les recherches en cours en milieux aquatiques portent principalement sur les poissons et les amphibiens (Figure 5), mais d’autres se développent rapidement sur une plus large gamme de communautés vivantes animales (invertébrés) et végétales (phanérogames, bryophytes, …).
Cette détection facilitée des espèces peut être considérée comme un atout dans l’amélioration de la gestion des invasions biologiques. Elle ne constitue toutefois que la première phase de la démarche du gestionnaire qui doit le conduire à la mise en place d’une gestion appropriée, dans laquelle la dimension quantitative des populations à gérer (nombre d’individus, biomasse…) reste un critère important des choix techniques qu’il a à faire. Le développement de méthodes utilisant l’ADNe pour caractériser autre chose que la présence d’une espèce dans un milieu donné semble donc encore largement à poursuivre pour apporter d’autres contributions efficaces aux besoins des gestionnaires.
Alain Dutartre (expert indépendant) et Emmanuelle Sarat (UICN France), avril 2016.
Fujiwara A., Matsuhashi S., Doi H., Yamamoto S., Minamoto T., 2016. Use of environmental DNA to survey the distribution of an invasive submerged plant in ponds. Freshwater Science. 35(2)
Kadono, Y., 2004. Alien aquatic plants naturalized in Japan: history and present status. Global Environmental Research 8: 163–169.
Scriver M., Marinich A., Wilson C., Freeland J., 2015. Development of species-specific environmental DNA (eDNA) markers for invasive aquatic plants. Aquatic Botany 122, 27–31
Eichmiller, J. J., P. G. Bajer, and P. W. Sorensen. 2014. The relationship between the distribution of common carp and their environmental DNA in a small lake. PLoS ONE 9: e112611.
Doi, H., K. Uchii, T. Takahara, S. Matsuhashi, H. Yamanaka, and T. Minamoto. 2015. Use of droplet digital PCR for estimation of fish abundance and biomass in environmental DNA surveys. PLoS ONE 10:e0122763.
Valentini A, Taberlet P, Miaud C et al. (2016) Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 24, 929–942.


